Section 4.3 Méthodes de réduction basées sur les distances
L'idée centrale des méthodes qu'on va présenter ici est qu’ on cherche un opérateur de réduction qui préserve au mieux les distances. En préservant les voisinages lors de la réduction on a cherché finalement à capturer les propriétés locales de la variété en préservant que les points proches reste proches lors de la réduction. Ici l'idée est de préserver les distances en deux coins lors de la réduction. En faisant cela, on espère capturer les propriétés globales de la variété. On commencera par une méthode linéaire (on cherche un hyperplan) qui sera utile par la suite
Subsection 4.3.1 Méthode du positionnement multidimensionnel
La méthode de positionnement multidimensionnel est une méthode qui ressemble à l'ACP en pratique. Cependant elle ne travaille pas sur la matrice des données, mais sur une matrice de similarité à travers une distance \(d\text{.}\) L'idée est de se munir d'une notion de distance qui mesure la similarité. Plus des données sont similaires/différentes plus la distance va être petite/grande. À partir de là on construira une réduction qui devra préserver ces similarités. On considère l'échantillon \(\mathcal{X}=(x_1,..,x_n)\) et la matrice de similarité/distance \(D\) qui normalement est une matrice dense de distance entre les données:
La matrice \(D\) peut être construite avec des distances issues de la norme \(p\) ou avec la distance de Manhattan. On se focalisera sur le cas \(p=2\) qui est plus simple. Avant de construire l'algorithme, on va rappeler des propriétés utiles à la suite.
Définition 4.14.
Soit une matrice \(D \in \mathcal{M}_{n,n}(\mathbb{R})\text{.}\) On dit qu'elle est une matrice de distance euclidienne si il existe \(d \gt 0\) et \(\mathcal{X}=(x_1,...,x_n)\in \mathbb{R}^d\) tel que
et la matrice de distance carrée euclidienne est définie par
Dans le cas l'ensemble \(\mathcal{X}\) est appelé ensemble de configuration.
Définition 4.15.
Soit l'ensemble \(\mathcal{X}=(x_1,...,x_n)\in \mathbb{R}^d\text{,}\) on appelle la matrice de Gram associée, la matrice \(G\in \mathcal{M}_{n,n}(\mathbb{R})\) tel que:
Cette matrice est symétrique positive. Si elle est de rang \(r\) il existe une décomposition de Cholesky \(G=X^tX\) avec \(X=(x_1,...,x_n)\mathcal{M}_{r,n}(\mathbb{R})\text{.}\)
On peut montrer qu'à l'inverse une matrice définie positive est une matrice de Gram pour un certain échantillon de point. On rappelle que
donc
On va montrer que cette relation est inchangée si on prend des données centrées. On définit le centre
si (comme on l'assume) les données vivent dans un hyperplan de dimension \(m\text{,}\) c'est aussi vrai pour le centre et donc le jeu de données centrées (\(\hat{x}_i=x_i-\bar{x})\)):
est aussi concentré sur un hyperplan parallèle au précédent. Soit la matrice de Gram associée aux données centrées:
avec la matrice le vecteur des éléments de \(\mathcal{X}\text{.}\) Un rapide calcul utilisant les propriétés du produit scalaire donne que :
Les matrices de Gram centrées seront importantes dans la suite. On introduit donc une dernière définition et des résultats.
Définition 4.16.
Soit \(A\) une matrice symétrique. On appelle la matrice centrée associé à \(A\) la matrice \(A^c=HAH\) avec \(H\) la matrice de centrement:
ou \(\mathbb{1}\)> le vecteur remplit de \(1\text{.}\) Une matrice est donc centrée si \(A=HAH\text{.}\)
Théorème 4.17.
Soit l'ensemble \(\mathcal{X}=(x_1,...,x_n)\in \mathbb{R}^d\text{.}\) Soit \(S\) la matrice de distance carrée euclidienne et \(G^c\) la matrice de Gram centrée associée. Dans ce cas on a
Preuve.
Par définition
On prend la relation entre \(G\) et \(D\text{:}\)
donc
et de la même façon:
On écrit ensuite les coefficients de \(S^c\text{:}\)
On utilise les relations trouvées précédemment pour obtenir
en utilisant (4.11).
Théorème 4.18.
Soit \(A\) une matrice symétrique.
\(A\) est une matrice de Gram si et seulement si elle est positive,
\(A\) est une matrice de Gram centrée si et seulement si elle est centrée,
A est une matrice de distance carrée euclidienne si et seulement si \(-\frac12 A^c\) est centrée positive,
Preuve.
Maintenant grâce à ces matrices de Gram, on va montrer qu'on peut effectuer une 1ère réduction de dimension puis que si la dimension obtenue est encore trop grande il est toujours possible d'en effectuer une deuxième en agissant de façon similaire à l'ACP. Pour commencer, on va introduire le principe du positionnement multidimensionnel. L'idée est de trouver une représentation en petite dimension qui préserve les distances/les voisinages. Cela revient à trouver \(\mathcal{Y}=(y_1,...y_n)\in \mathbb{R}^m\) tel qu’ une matrice de distance associée à \(\mathcal{Y}\) noté \(D_y\) soit proche de \(D\) la matrice distance centrée associée à \(\mathcal{X}\) dans \(\mathbb{R}^d\text{.}\) Cela équivaut a chercher \(\mathcal{Y}\)> tel que
On va d'abord montrer qu'une première réduction est rapidement possible. Soit \(S\) la matrice distance carrée euclidienne associée à \(D\text{.}\) On prend
la matrice de Gram centrée. On suppose que le rang de \(G^c\) est \(r\text{.}\) Par conséquent puisqu'il s'agit d'une matrice symétrique positive il existe une décomposition de Cholesky \(G^c=X^tX\) avec \(X\) (configuration exacte) la matrice de vecteur
Cela revient à dire que :
Ce résultat veut dire que si le rang de \(G_c\) est \(r\) (la configuration intrasecque de \(D\)) les données de l'échantillon centré sont en pratique dans \(\mathbb{R}^r\text{.}\) Si on s'arrête la on a trouver l'hyperplan de dimension \(r\) sur lequel les données sont distribuées. Cependant est possible/probable que \(r\) soit encore trop important. On va donc chercher l'hyperplan de dimension \(m \lt \lt r\) le meilleur possible. Pour cela on introduit la fonctionnelle:
avec \(T\) le projecteur orthogonal de \(\mathbb{R}^r\) dans un hyperplan de \(\mathbb{R}^k\text{.}\) On a la solution
De la même façon que, comme pour l'ACP, la solution sera donnée par une SVD.
Théorème 4.19.
Soit \(\mathcal{Z}\) la configuration exacte de la matrice \(D\) de l'échantillon centré \(\hat{\mathcal{X}}\) avec la SVD de \(Z\) donnée par
avec \(\Sigma_r=diag(\sigma_1,...,\sigma_r)\text{.}\) Pour \(m \lt \lt d\text{,}\) soit \(V_d=[v_1, ...v_d]\) la solution du problème de minimisation est donnée par \(Y=V_d^{t} Z\) avec
Preuve.
On définit \(B \in \mathcal{M}_{r,m}\) la matrice de la base orthonormal de \(S_m\) sous-espace de \(\mathbf{R}^r\text{.}\) On a donc \(B B^t\) le projecteur sur \(S_m\text{.}\) On rappelle que
donc
(??) et
donc
car \(B B^{t}\) est une matrice unitaire. On voit que le problème de minimisation s'écrit: On cherche \(B_*\in \mathcal{M}_{r,m}\) tel que \(B_*^t\mathcal{Z}\) est solution du problème
ce qui équivaut à
TOOOOODOOO: finish
Lemme 4.20.
Le vecteur réduit \(Y=(y_1,..,y_n)\) avec chaque composante dans \(\mathbb{R}^k\) est donné par
avec \(U_k\) les \(k\) 1er vecteurs propres de \(G_c\) et \(\Lambda_k\) les \(k\) 1ères valeurs propres de \(G_c\)
Preuve.
En effet:
de plus \(Y_r= V^t Z\) ou \(V^t X = V^t V \sigma_r U = \sigma_r U^t\text{.}\) Lorqu'on on fait une décomposition spectrale de \(G_c\) on obtient \(U \Lambda U^t\) et donc on voit qu'on peut obtenir la matrice \(Y_r\) par
avec \(U\) les vecteurs propres de \(G_c\) et \(\lambda\) les valeurs propres. Par conséquent
avec \(U_k\) les \(k\) 1ers vecteurs propres de \(G_c\) et \(\Lambda_k\) les \(k\) 1ère valeurs propres.
Algorithm 4.21. Algorithme MDS.
On connait en entrée une matrice de distance,
On calcule la matrice de Gram centrée \(G=-\frac12 H D^2 H\text{,}\)
On fait une décomposition spectrale de \(G= U \Lambda G^t\) avec \(U=[u_1,...u_n]\) et \(Diag([\lambda_1,....,\lambda_n])\)
On se restreint à avec \(U_d=[u_1,...u_d]\) et \([\lambda_1,....,\lambda_d]\) et on obtient la projection \(Y= \sqrt{\Lambda_k}U_k^t\)
L 'algorithme est en pratique proche de l'algorithme d'ACP mais travaille sur une matrice différente obtenue à partir de la matrice des distance centrée. On peux choisir une autre distance que la distance euclidienne.
Subsection 4.3.2 Méthode ISOMAP
La méthode ISOMAP comme les méthodes LLE et LTSA va utiliser des outils de géométrie différentielle afin de capturer la variété de basse dimension sous-acjente aux données. Cependant elle n'est pas basée sur la notion de voinisage, mais basée sur la notion de distance. On part de la même idée que la méthode MDS. Le plongement doit préserver les distances entre les points. On rappelle que si on se donne une carte locale:
elle satisfait que \(d_e(\phi_{x_1}(x_1),\phi_{x_2}(x_2))=d_{\mathcal{M}}(x_1,x_2)\) ce qui équivaut notamment à
C'est à dire que la distance géodésique (la distance intraséque à la variété entre deux points est la même que la distance entre deux coordonnées locales dans \(\mathbb{R}^m\text{.}\) En construisant donc une réduction qui préserve ces distances on peut donc espérer trouver une \(carte locale\) de la variété en basse dimension et donc un plongement qui capture la variété. Cependant on n’a pas a priori accès à la distance géodésique. Une première solution serait d'utiliser la distance euclidienne comme dans l'algorithme MDS pour approcher cette distance. Prenons un exemple. On se place dans \(\mathbb{R}^2\) avec des points distribués selon une variété de dimension 1. On compare les voisinages au sens des distances de \(\mathbb{R}^2\) et géodésiques. L'idée centrale d'ISOMAP est de trouver un plongement qui préserve des distances géodésiques approchées associées à la variété sous-acjente aux données en appliquant un algorithme de type MDS à la matrice des distances géodésiques.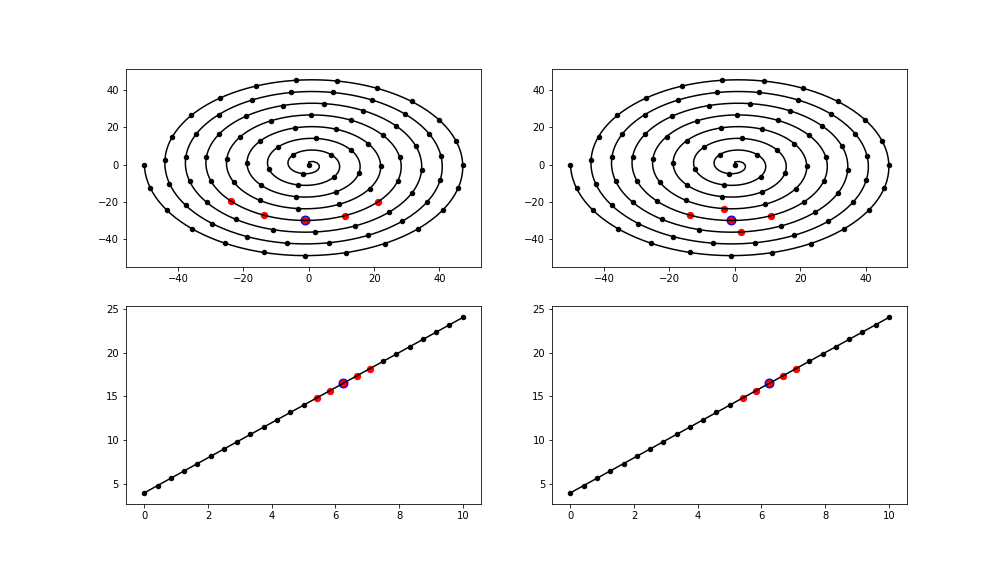
Idée d'ISOMAP.
Pour estimer cette distance l'idée est la suivante:
Construire un graphe issu des données reliant les poids les plus proches qui a pour but "d'approcher la variété".
Approcher la distance géodésique par la distance des graphes: le plus court chemin. Construire la matrice de similarité avec.
Pour construire le graphe, on utilise l'algorithme de plus proches voisins introduit précédemment. On parle de graphe des plus proches voisins.
Définition 4.23.
Soit un nuage de point \((x_1,....x_n)\in \mathbf{R}^d\text{.}\) Le graphe des plus proches voisins est le graphe à poids \(\mathcal{G}(V,E)\) avec \(V=(x_1,....x_n)\) et \(E\) l'ensemble des arêtes reliant tous les points tels que les poids sont donnés par
avec \(KNN(x)\) l'ensemble des voisins de \(x\) au sens de l'algorithme des \(k\) plus proches voisins.
Une fois un tel graphe construit on peut donc approcher la distance géodésique entre deux points à l'aide d'algorithmes du plus cours chemin. La convergence entre la distance du plus court chemin sur le graphe vers la distance géodésique est asymptotique avec le nombre de points (voir sous sous-section suivante). D'abord on comprend immédiatement que si le nombre de points est trop faible on ne pourra pas capturer les distances géodésiques. Mais le paramètre \(K\) de l'algorithme des \(K\) voisins joue aussi un rôle important. En effet si \(K\) est trop faible il peut avoir des zones/noeuds du graphe non connecté entre elles (essentiel pour le calcul du plus court chemin).
Comparaison géodésique interactive sur la spirale variation nombre de point et K pour peu de points.
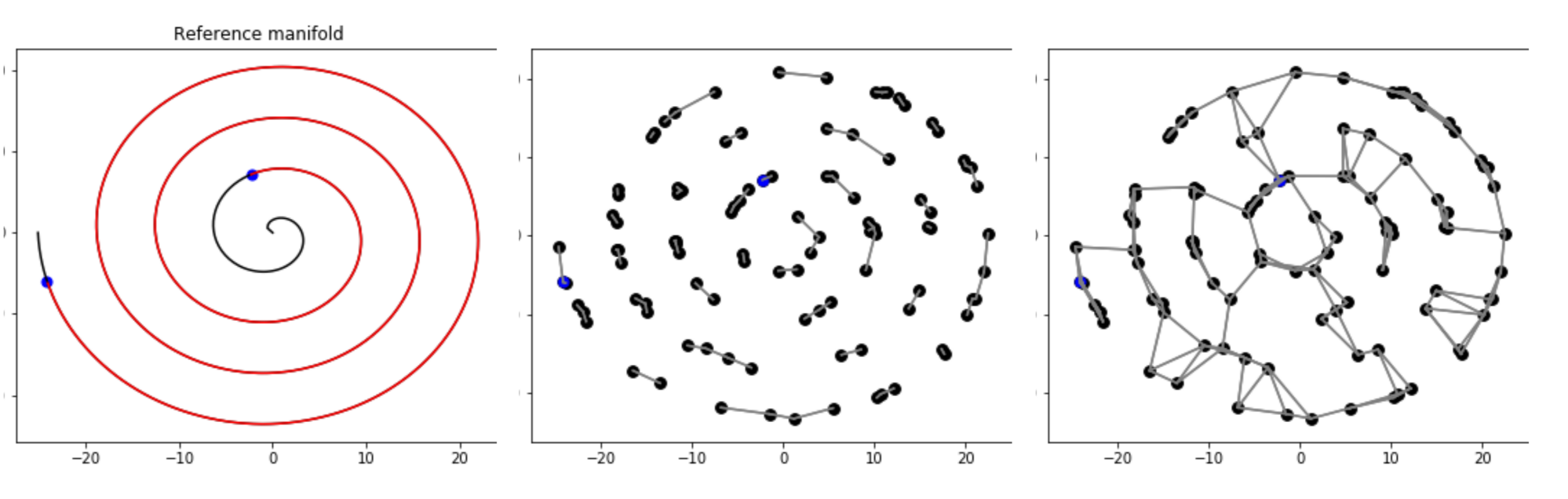
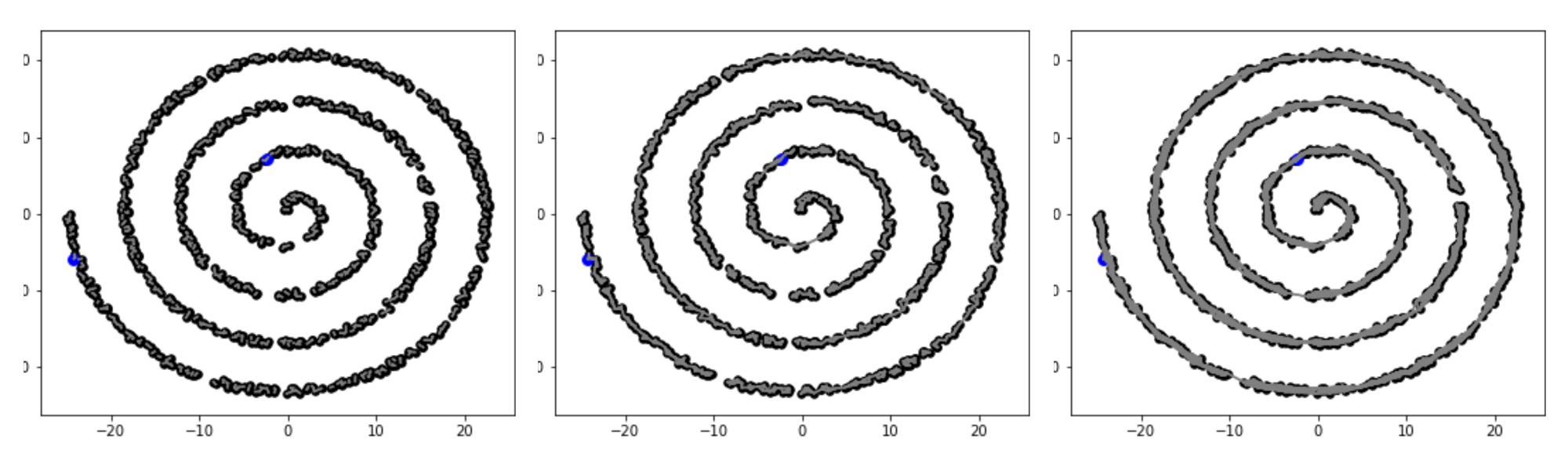
Sur la figure Figure 4.24 on voit vite que si \(K\) est trop petit des portions du graphe ne sont pas connectées entre elles. On voit aussi que plus le nombre de points \(N\) est grand plus \(K\) doit être pris grand pour que tous les noeuds soient connectés. Sur l'exemple, on voit que pour \(N=100\text{,}\) \(K=4\) suffit alors que pour \(N=1000\text{,}\) le choix \(K=10\) ne suffit pas tout à fait. À l'inverse on voit aussi qu'on ne peut pas prendre \(K\) arbitrairement grand si on veut correctement capturer la distance géodésique. On voit sur la figure Figure 4.25 que pour \(K=12\) on capture bien la courbe géodésique alors que dans les deux autres cas on le capture pas. On voit que pour \(K\) trop grand le graphe approche de plus en plus la surface sur lequel il y a les points et non la variété 1D. Il s'agit d'un problème à \(N\) fixé il disparaît quand le nombre de points tend vers l'infini.
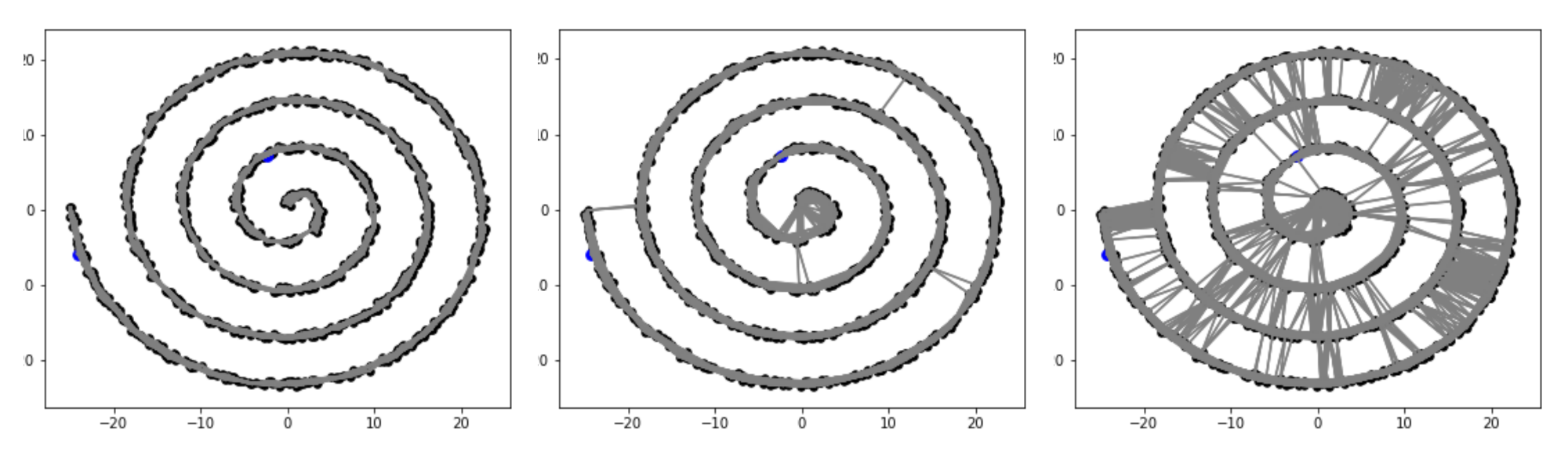
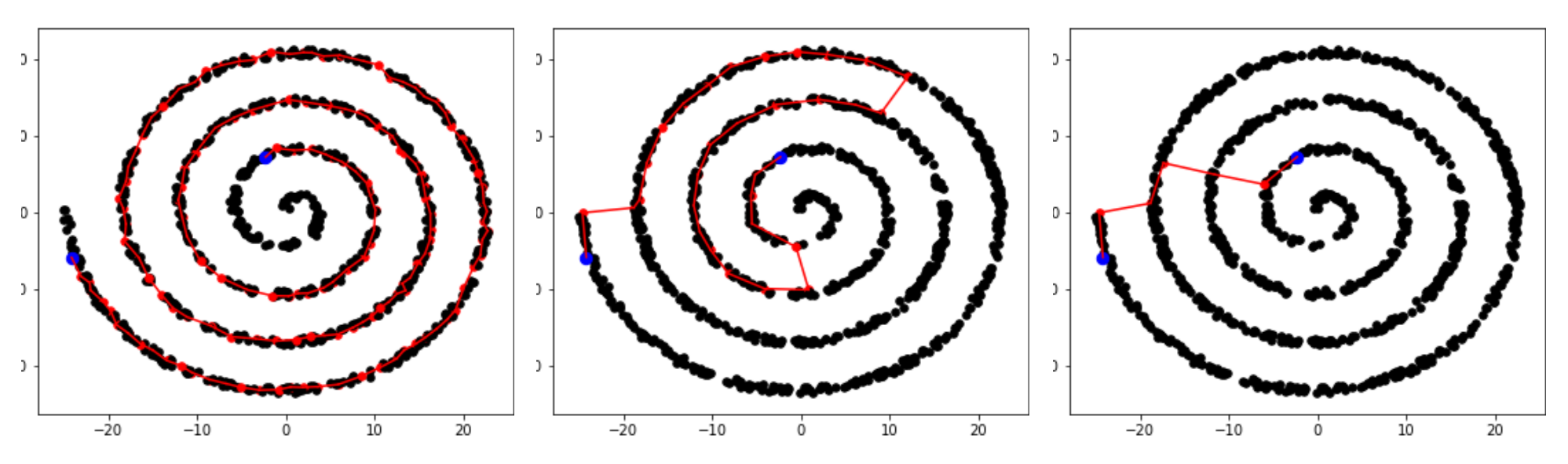
Ces constatations sur le choix de \(K\) sont validés par un théorème de convergence que l'ont va introduire par la suite. Une fois les distances géodésiques calculées on peut appliquer la méthode MDS. Pour cela on:
On calcule la matrice \(S\) tel que \(S_{ij}= d_g(x_i,x_j)\)
On calcule ma matrice centrée associée: \(S^c = H S H\)
On obtient la matrice de Gram pour l'échantillon \((y_1,..y_n)\) donné par \(G^c=-\frac12S^c\) et on applique une ACP a cette matrice. On parle du noyau "ISOMAP".
Si le nombre de points n'est pas assez important la distance géodésique peut être mal approchée et la matrice \(D^c\) va mal approcher la distance géodésique et donc la distance euclidienne dans l'espace réduit (puisqu'on cherche un plongement dont la distance euclidienne en basse dimension correspond à la distance géodésique). Par conséquent la matrice \(G^c\) peut ne pas s'approcher d'une matrice de Gram et perdre son caractère définie potisive. Dans ce cas des corrections existent qu'on ne détaillera pas. Au final on peut résumé la méthode avec l'algorithme suivant:
Algorithm 4.26. Algorithme ISOMAP.
On calcule le graphe des plus proches voisins,
\(\forall i,j\lt n\text{:}\)
on calcul la distance du plus court chemin \(d_c(x_i,x_j)\) afin de construire la matrice de similarité \(D_g\)
on applique l'algorithme de positionnement multidimensionnel à la matrice \(D_g\) obtenue.
Maintenant on va montrer les résultats numériques qui valident l'approche. On en profitera pour discuter certaines limites.
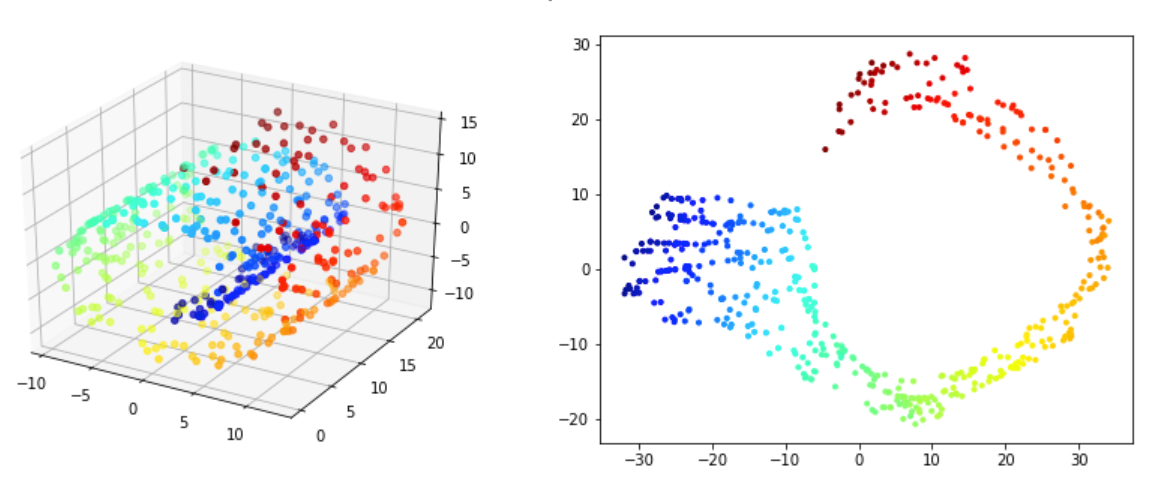
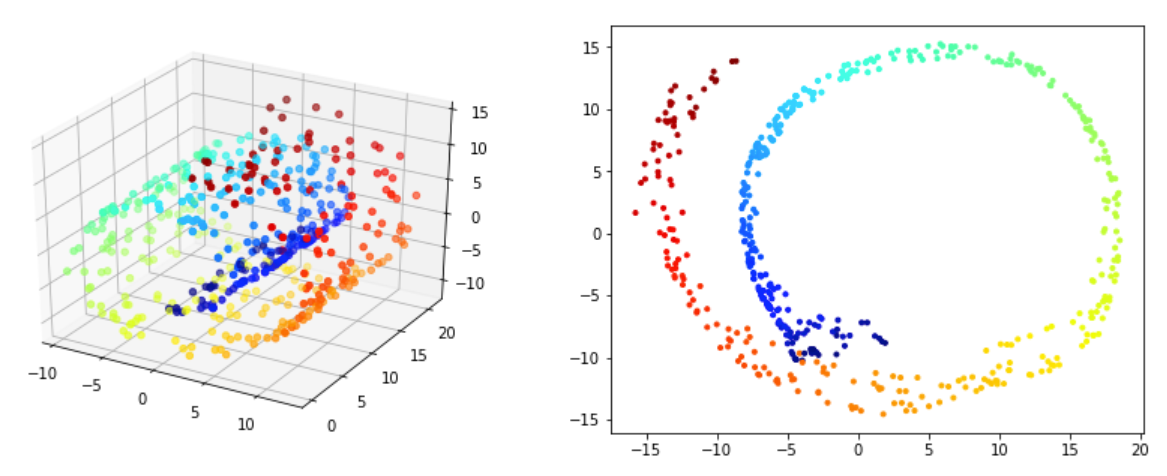
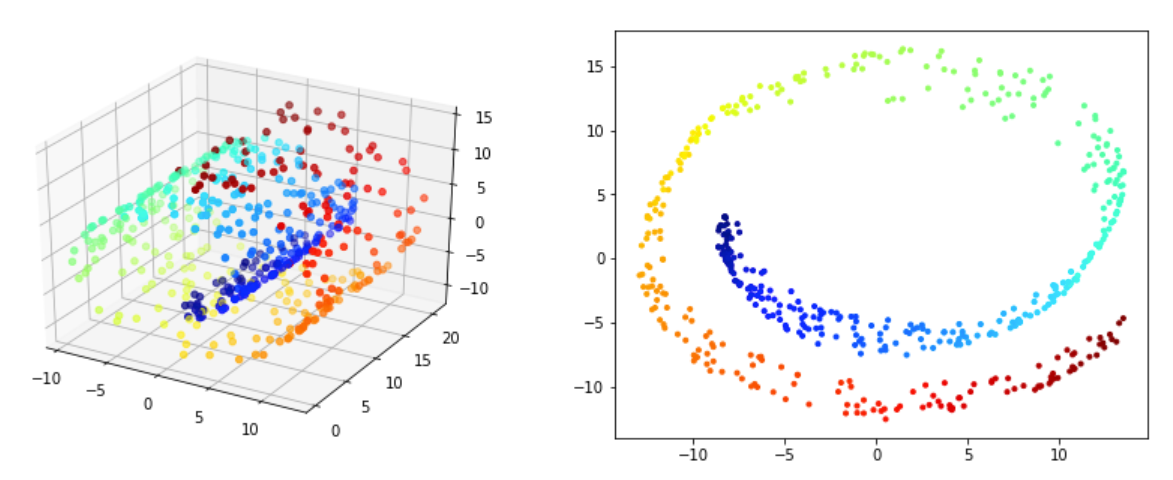
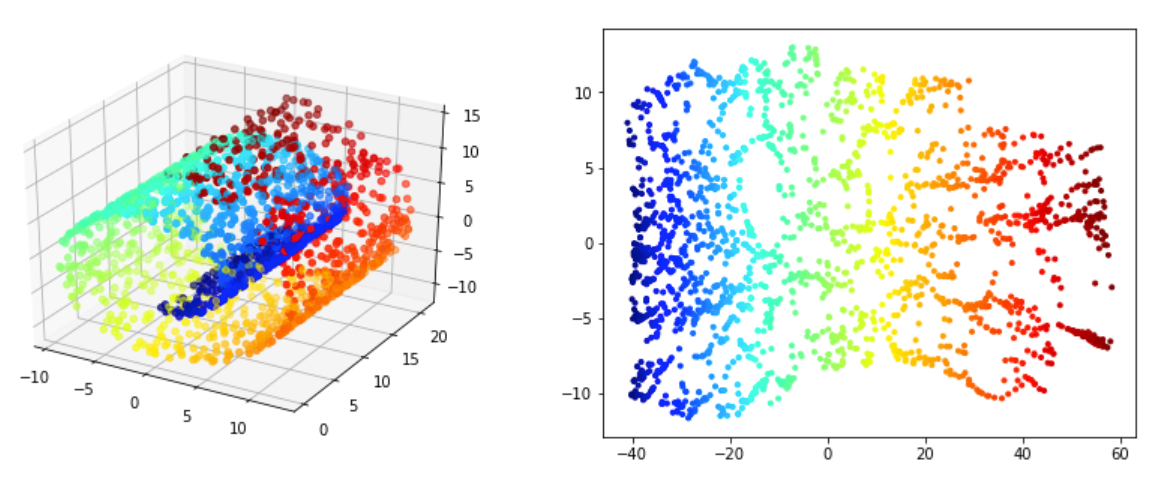
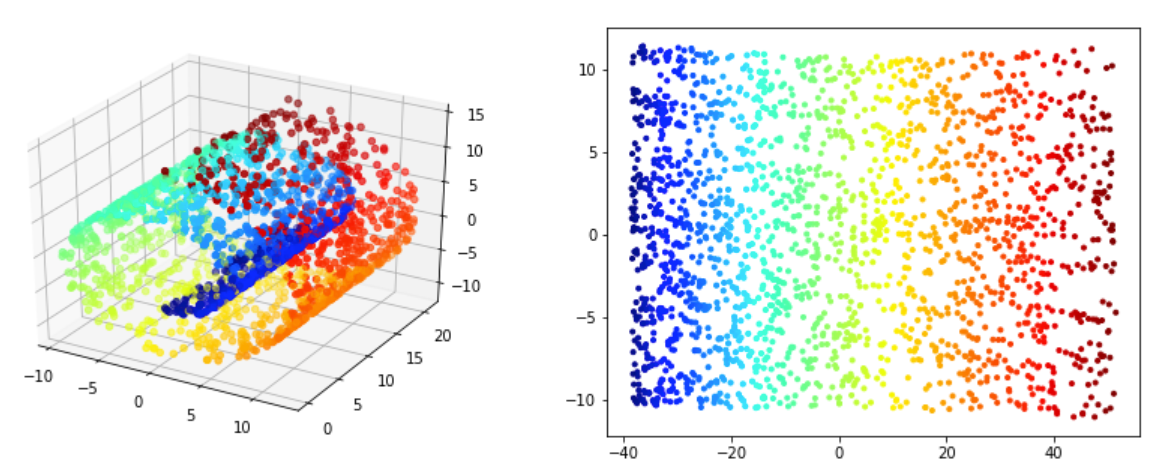

Sur les figures Figure 4.27 - Figure 4.28 on valide la méthode ISOMAP. On souhaite que les voisinnages soit préservés dans la projection 2D. Pour 500 points Figure 4.27 on voit qu'on arrive a déplier partiellement la variété mais les voisinages ne préservés qu'a des niveaux trè locales (les points rouge sont par example assez proches des bleus ce qui n'est pas vrai au sens de la variété). Le meilleur résultat semble être obtenu pour \(n_{voisins}=5\text{.}\) Pour 2000 points Figure 4.28 on obtient de très bon résultats pour \(n_{voisins}=20\text{.}\) Cela confirme que si on a pas assez de points les résultats ne seront pas assez bon. Si on a un nombre suffisement de points il faut bien choisir le nombre de voisins. un nombre trop grand ou trop petit dégrade les résultats.
Subsubsection 4.3.2.1 Résultat de convergence
On va maintenant introduire qui montre que la distance au sens du plus court chemin sur un graphe des plus proches voisins converge vers la distance géodésique de la variété régulière auxquelles appartiennent les sommets du graphe.
En reconstruction
Subsection 4.3.3 Méthode PTU
../../../../CodeFrench.html#SciML2../../../../CodeFrench.html#SciML2